1.1 El descubrimiento de los virus gigantes
Tres descubrimientos recientes han puesto en duda las 5 respuestas del modelo centrado en el virus como genes escapados en vehículos llamados viriones: (1) El descubrimiento de virus gigantes, incluso tan grandes como la bacteria E. coli (~2um), el caso del enigmático Pithovirus (~1.5um) (Figura 2). El cual cuenta con una complejidad genética única e incluso con un tamaño mayor al de bacterias parásitas obligadas (Mycoplasma pneumoniae 0.8Mb vs Pandoravirus 2.5Mb). (II) La existencia de virus que parasitan otros virus, el caso de los viriofagos, quienes no solo dependen de otros virus para replicarse (como los virus satélite), sino que van más allá y parasitan la fábrica viral o el centro de replicación del virus del cual dependen.
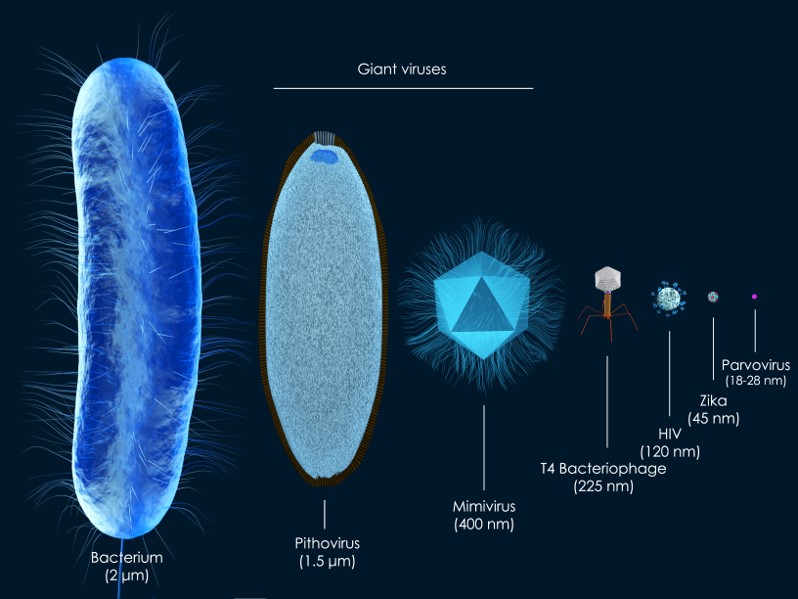
Figura 1.2: Comparación del tamaño de la estrucutra dispersiva de diversos virus frente a la bacteria E. coli, creditos de la imagen a Meletios Verras stock illustration ID: 720441124
- La mayor dificultad al modelo de los virus como genes escapados en viriones es la presencia de genes exclusivos a los virus, como los genes que codifican las proteínas que forman las cápsides de los viriones o los responsables de la replicación del genoma viral, no obstante un descubrimiento fascinante en esta área ha demostrado que, aunque en su secuencia de ADN en virus de los 3 diferentes dominios (clásicos) de la vida (Bacteria, Arqueas y Eucariotas), NO hay evidencia de un único origen (o un ancestro viral común), esta tarea si es posible desde el punto de vista estructural de las proteínas de la cápside, como lo evidencia el plegamiento de los monómeros de la cápside de: Chlorella virus que infecta eucariotas, El bacteriófago PrD1 y el virus de arqueas termófilas STIV (Figura 3). El genoma de todos los anteriores virus es a base de ADN y de cadena doble, no obstante el dominio estructural llamado “jelly-roll fold” (o pliegue de gelatina) se encuentra en virus de cadena sencilla de ARN y ADN también, sugiriendo una historia evolutiva común (5) e independiente de la vida celular (4).
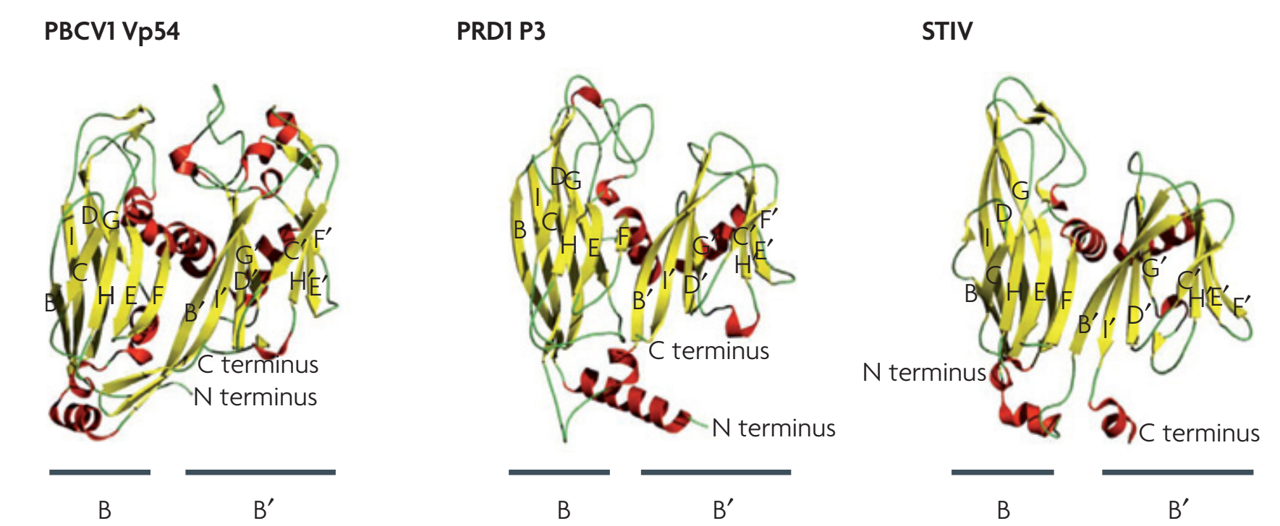
Figura 1.3: Desde el punto de vista estructural de las proteínas de la cápside los virus parecen compartir un ancestro común, como lo evidencia el plegamiento de los monómeros de la cápside de: Chlorella virus que infecta eucariotas, El bacteriófago PrD1 y el virus de arqueas termófilas STIV, tomado de [5]